R语言:microeco:一个用于微生物群落生态学数据挖掘的R包,第四:trans
trans_beta class:利用trans_beta类可以变换和绘制beta分集的距离矩阵。该类中涉及到beta多样性的分析主要包括排序、群距、聚类和方差分析。我们首先使用PCoA显示排序。
> dataset$cal_betadiv() The result is stored in object$beta_diversity ... > t1 t1$cal_ordination(ordination = "PCoA") The ordination result is stored in object$res_ordination ... > tmp t2 t2$cal_diff(group = "Group", method = "anova") The result is stored in object$res_diff ... Warning message: 程辑包‘agricolae’是用R版本4.3.3 来建造的 #这里需要安装agricolae包,直接install就行。 > t2 trans_env object: Env table have 2 variables: PCo1,PCo2
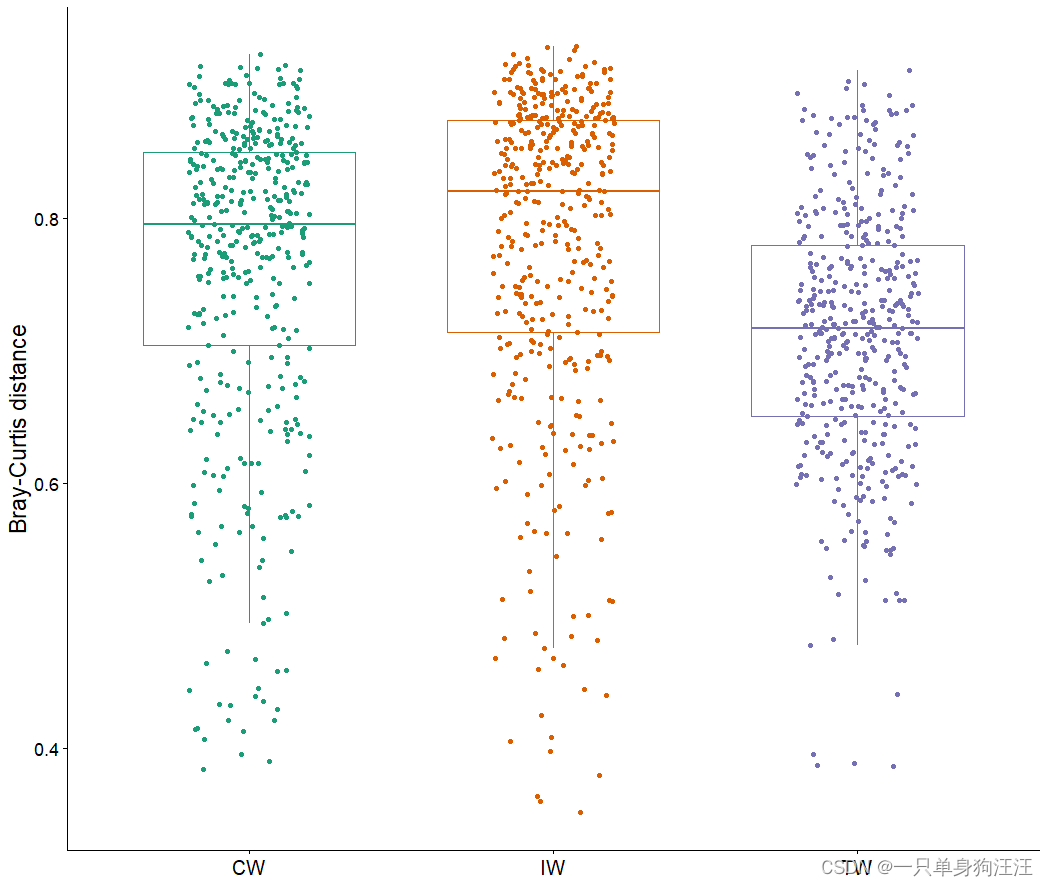
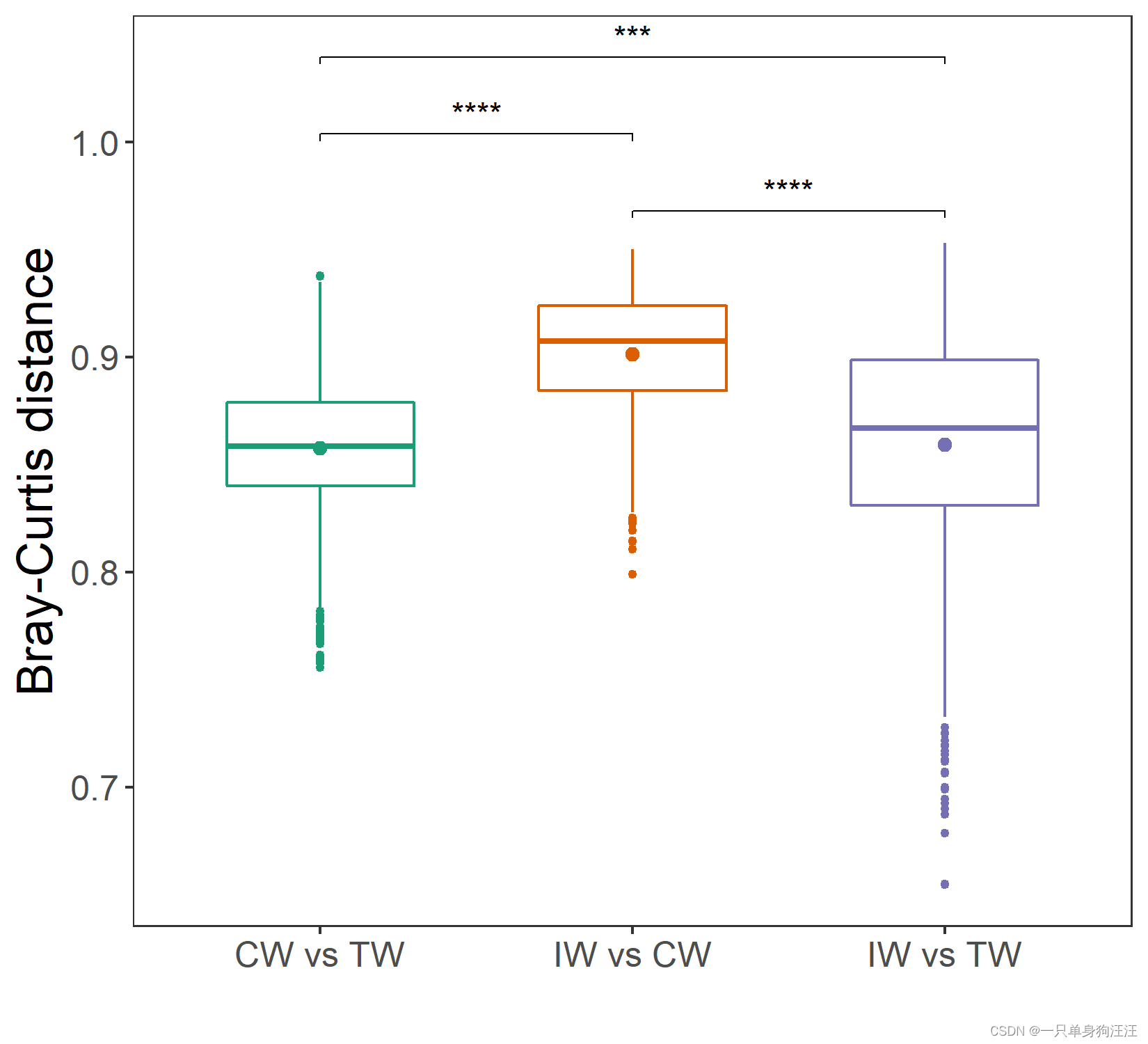
> p1 t1$cal_group_distance()
> t1$plot_group_distance(distance_pair_stat = TRUE)
#这里应该会有差异比较的,但是却没有。示例如下,不知道有什么问题,等到有需求我会解决这个问题。

(图片来源网络,侵删)
#计算和绘制组之间的样本距离
> t1$cal_group_distance(within_group = FALSE)
> t1$plot_group_distance(distance_pair_stat = TRUE)
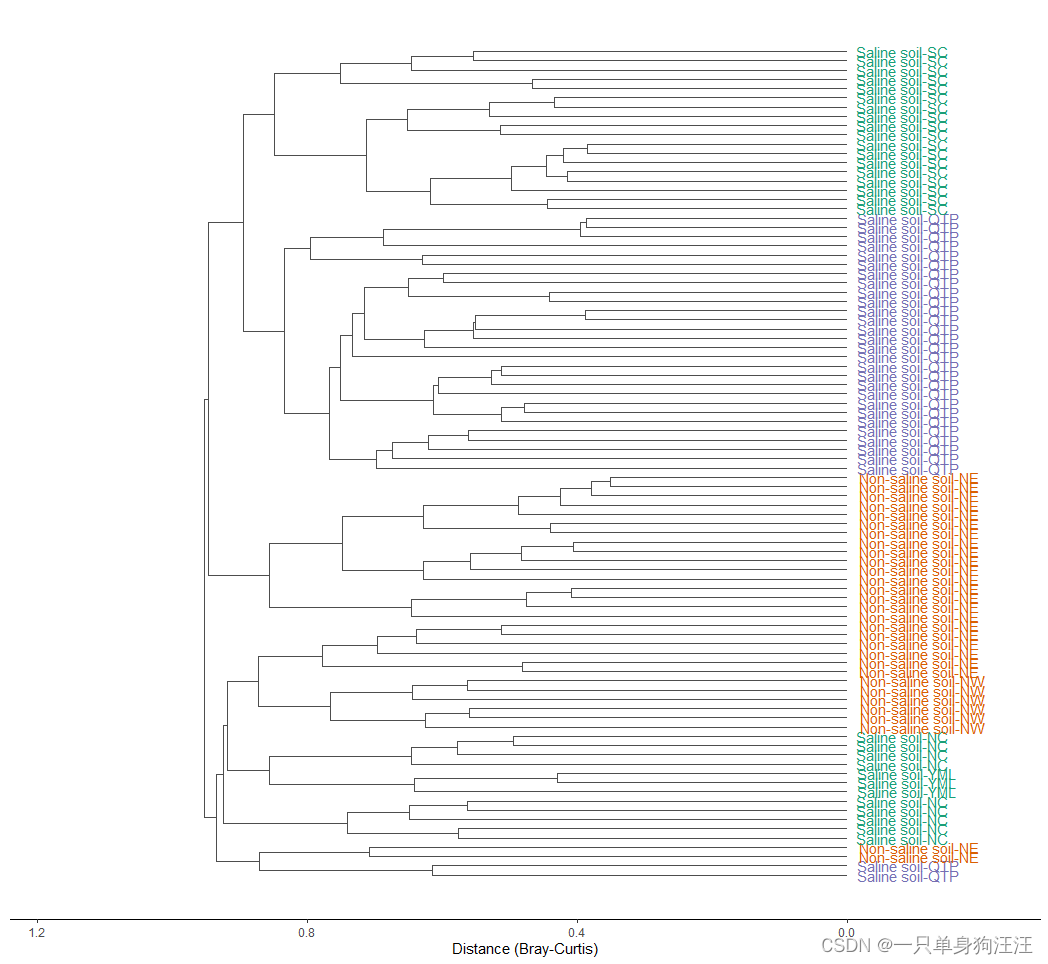
# 聚类图也是一种常用的方法。

(图片来源网络,侵删)
> t1$plot_clustering(group = "Group", replace_name = c("Saline", "Type"))
#perMANOVA常用于组间距离的差异检验。
> t1$cal_manova(cal_manova_all = TRUE)
The result is stored in object$res_manova ...
> t1$res_manova
Permutation test for adonis under reduced model
Terms added sequentially (first to last)
Permutation: free
Number of permutations: 999
adonis2(formula = use_formula, data = metadata, cal_manova_all = TRUE)
Df SumOfSqs R2 F Pr(>F)
Group 2 6.1207 0.19553 10.573 0.001 ***
Residual 87 25.1822 0.80447
Total 89 31.3029 1.00000
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
## manova表示每一对组
> t1$cal_manova(cal_manova_paired = TRUE)
The result is stored in object$res_manova ...
> t1$res_manova
Permutation test for adonis under reduced model
Terms added sequentially (first to last)
Permutation: free
Number of permutations: 999
adonis2(formula = use_formula, data = metadata, cal_manova_paired = TRUE)
Df SumOfSqs R2 F Pr(>F)
Group 2 6.1207 0.19553 10.573 0.001 ***
Residual 87 25.1822 0.80447
Total 89 31.3029 1.00000
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
# manova用于指定组集:这里是“group + Type”
> t1$cal_manova(cal_manova_set = "Group + Type")
The result is stored in object$res_manova ...
> t1$res_manova
Permutation test for adonis under reduced model
Terms added sequentially (first to last)
Permutation: free
Number of permutations: 999
adonis2(formula = use_formula, data = metadata, cal_manova_set = "Group + Type")
Df SumOfSqs R2 F Pr(>F)
Group 2 6.1207 0.19553 10.573 0.001 ***
Residual 87 25.1822 0.80447
Total 89 31.3029 1.00000
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
这个包的改动有些大,以前的代码不能直接跑了。我发觉,学习这个包还是要基于需求啊,否则会很快忘掉。以前我使用vegan包分析+ggplot2包做PCOA分析,等把这个包分享完了,我会再分享其它的R语言。
文章版权声明:除非注明,否则均为主机测评原创文章,转载或复制请以超链接形式并注明出处。






还没有评论,来说两句吧...